15 データハンドリング [拡張]
これまでデータの一部(subset)を抽出したり、要約する方法について説明しましたが、本章はデータの拡張について解説します。具体的にはデータ内の変数に基づき、指定された計算を行った結果を新しい列として追加する、または既存の変数を更新する方法です。そして2つ以上のデータセットを1つに結合する方法についても解説します。今回も前章と同じデータを使用します。
データの詳細については第13.3章を参照してください。
15.1 変数の計算
15.1.1 mutate()関数の使い方
データフレーム(ティブル)内の変数を用いて計算を行い、その結果を新しい列として格納するmutate()関数について紹介します。まず、mutate()関数の書き方からです。
これは何らかの処理を行い、その結果を新しい変数としてデータフレームに追加することを意味します。新しく出来た変数は、基本的に最後の列になります。ここでは分単位であるWalkを時間単位に変換したWalk_Hour変数を作成するとします。処理内容はWalk / 60です。最後に、都府県名、店舗名、徒歩距離 (分)、徒歩距離 (時間)のみを残し、遠い順にソートします。
df |>
filter(!is.na(Walk)) |>
mutate(Walk_Hour = Walk / 60) |>
select(Pref, Name, Walk, Walk_Hour) |>
arrange(desc(Walk_Hour))# A tibble: 5,375 × 4
Pref Name Walk Walk_Hour
<chr> <chr> <dbl> <dbl>
1 埼玉県 札幌ラーメン どさん子 小鹿野店 116 1.93
2 神奈川県 札幌ラーメン どさん子 中津店 73 1.22
3 千葉県 札幌ラーメン どさん子 佐原51号店 59 0.983
4 神奈川県 札幌ラーメン どさん子 山際店 50 0.833
5 千葉県 札幌ラーメン どさん子 関宿店 49 0.817
6 兵庫県 濃厚醤油 中華そば いせや 玉津店 43 0.717
7 大阪府 河童ラーメン本舗 岸和田店 38 0.633
8 埼玉県 ラーメン山岡家 上尾店 35 0.583
9 兵庫県 濃厚醤油 中華そば いせや 大蔵谷店 35 0.583
10 大阪府 河童ラーメン本舗 松原店 31 0.517
# ℹ 5,365 more rowsmutate()は3行目に登場しますが、これはWalkを60に割った結果をWalk_Hourとしてデータフレームの最後の列として格納することを意味します。もし、最後の列でなく、ある変数の前、または後にしたい場合は、.beforeまたは.after引数を追加します。これはselect()関数の.beforeと.afterと同じ使い方です。たとえば、新しく出来たWalk_HourをIDとNameの間に入れたい場合は
のようにコードを修正します。
むろん、変数間同士の計算も可能です。たとえば、以下のようなdf2があり、1店舗当たりの平均口コミ数を計算し、ScoreN_Meanという変数名でScoreN_Sumの後に格納うするとします。この場合、ScoreN_Sum変数をNで割るだけです。
# A tibble: 9 × 6
Pref Budget_Mean ScoreN_Sum ScoreN_Mean Score_Mean N
<chr> <dbl> <dbl> <dbl> <dbl> <int>
1 京都府 1399. 216 0.522 3.68 414
2 兵庫県 1197. 230 0.389 3.54 591
3 千葉県 1124. 259 0.259 3.72 1000
4 和歌山県 1252 83 0.593 3.97 140
5 埼玉県 1147. 278 0.278 3.64 1000
6 大阪府 1203. 516 0.516 3.77 1000
7 奈良県 1169. 45 0.306 3.85 147
8 東京都 1283. 1165 1.16 3.67 1000
9 神奈川県 1239. 587 0.587 3.53 1000このように、データ内の変数を用いた計算結果を新しい列として追加する場合は、mutate()が便利です。これをmutate()を使わずに処理する場合、以下のようなコードになりますが、可読性が相対的に低いことが分かります。
むろんですが、計算には+や/のような演算子だけでなく、関数を使うことも可能です。たとえば、Budgetが1000円未満なら"Cheap"、1000円以上なら"Expensive"と示す変数Budget2を作成する場合はifelse()関数が使えます。
df |>
mutate(Budget2 = ifelse(Budget < 1000, "Cheap", "Expensive")) |>
filter(!is.na(Budget2)) |> # Budget2が欠損した店舗を除外
group_by(Pref, Budget2) |> # PrefとBudget2でグループ化
summarise(N = n(), # 店舗数を表示
.groups = "drop")# A tibble: 18 × 3
Pref Budget2 N
<chr> <chr> <int>
1 京都府 Cheap 22
2 京都府 Expensive 28
3 兵庫県 Cheap 39
4 兵庫県 Expensive 27
5 千葉県 Cheap 64
6 千葉県 Expensive 72
7 和歌山県 Cheap 10
8 和歌山県 Expensive 5
9 埼玉県 Cheap 37
10 埼玉県 Expensive 45
11 大阪府 Cheap 104
12 大阪府 Expensive 115
13 奈良県 Cheap 11
14 奈良県 Expensive 10
15 東京都 Cheap 206
16 東京都 Expensive 236
17 神奈川県 Cheap 66
18 神奈川県 Expensive 54これは各都府県ごとの予算1000円未満の店と以上の店の店舗数をまとめた表となります。もし、500円未満なら"Cheap"、500円以上~1000円未満なら"Reasonable"、1000円以上なら"Expensive"になるBudget3変数を作るにはどうすればよいでしょうか。第11章で紹介しましたifelse()を重ねることも出来ますが、ここではcase_when()関数が便利です。まずは、ifelse()を使ったコードは以下の通りです。
case_when()を使うと以下のような書き方になります。
# case_when()を使う場合
df |>
mutate(Budget3 = case_when(Budget < 500 ~ "Cheap",
Budget >= 500 & Budget < 1000 ~ "Reasonable",
Budget >= 1000 ~ "Expensive"),
# 新しく出来た変数をfactor型にその場で変換することも可能
Budget3 = factor(Budget3,
levels = c("Cheap", "Reasonable", "Expensive"))) |>
filter(!is.na(Budget3)) |>
group_by(Pref, Budget3) |>
summarise(N = n(),
.groups = "drop")書く手間の観点ではcase_when()はifelse()と大きく違いはないかも知れませんが、コードが非常に読みやすくなっています。case_when()関数の書き方は以下の通りです。
似たような機能をする関数としてrecode()関数があります1。これは変数の値を単純に置換したい場合に便利な関数です。たとえば、都府県名をローマ字に変換するケースを考えてみましょう。
# recode()を使う場合
df2 |>
mutate(Pref2 = recode(Pref,
"東京都" = "Tokyo",
"神奈川県" = "Kanagawa",
"千葉県" = "Chiba",
"埼玉県" = "Saitama",
"大阪府" = "Osaka",
"京都府" = "Kyoto",
"兵庫県" = "Hyogo",
"奈良県" = "Nara",
"和歌山県" = "Wakayama",
.default = "NA"))# A tibble: 9 × 6
Pref Budget_Mean ScoreN_Sum Score_Mean N Pref2
<chr> <dbl> <dbl> <dbl> <int> <chr>
1 京都府 1399. 216 3.68 414 Kyoto
2 兵庫県 1197. 230 3.54 591 Hyogo
3 千葉県 1124. 259 3.72 1000 Chiba
4 和歌山県 1252 83 3.97 140 Wakayama
5 埼玉県 1147. 278 3.64 1000 Saitama
6 大阪府 1203. 516 3.77 1000 Osaka
7 奈良県 1169. 45 3.85 147 Nara
8 東京都 1283. 1165 3.67 1000 Tokyo
9 神奈川県 1239. 587 3.53 1000 Kanagawa使い方は非常に直感的です。
最後の.default引数は、もし該当する値がない場合に返す値を意味し、長さ1のベクトルを指定します。もし、指定しない場合はNAが表示されます。また、ここには紹介しておりませんでしたが、.missing引数もあり、これは欠損値の場合に返す値を意味します。
もう一つ注意すべきところは、今回はcharacter型変数をcharacter型へ変換したため、「"東京都" = "Tokyo"」のような書き方をしました。しかし、numeric型からcharacter型に変換する場合は数字の部分を`で囲む必要があります。たとえば、「`1` = "Tokyo"」といった形式です。ただし、character型からnumeric型への場合は「"東京都" = 1」で構いません。
recode()は値をまとめる際にも便利です。たとえば、EastJapanという変数を作成し、関東なら1を、それ以外なら0を付けるとします。そして、これはPref変数の後に位置づけます。
# 都府県を関東か否かでまとめる
df2 |>
mutate(EastJapan = recode(Pref,
"東京都" = 1,
"神奈川県" = 1,
"千葉県" = 1,
"埼玉県" = 1,
"大阪府" = 0,
"京都府" = 0,
"兵庫県" = 0,
"奈良県" = 0,
"和歌山県" = 0,
.default = 0),
.after = Pref)# A tibble: 9 × 6
Pref EastJapan Budget_Mean ScoreN_Sum Score_Mean N
<chr> <dbl> <dbl> <dbl> <dbl> <int>
1 京都府 0 1399. 216 3.68 414
2 兵庫県 0 1197. 230 3.54 591
3 千葉県 1 1124. 259 3.72 1000
4 和歌山県 0 1252 83 3.97 140
5 埼玉県 1 1147. 278 3.64 1000
6 大阪府 0 1203. 516 3.77 1000
7 奈良県 0 1169. 45 3.85 147
8 東京都 1 1283. 1165 3.67 1000
9 神奈川県 1 1239. 587 3.53 1000ただし、関東以外は全て0になるため、以下のように省略することも可能です。
# .default引数を指定する場合
df3 <- df2 |>
mutate(EastJapan = recode(Pref,
"東京都" = 1,
"神奈川県" = 1,
"千葉県" = 1,
"埼玉県" = 1,
.default = 0),
.after = Pref)
df3# A tibble: 9 × 6
Pref EastJapan Budget_Mean ScoreN_Sum Score_Mean N
<chr> <dbl> <dbl> <dbl> <dbl> <int>
1 京都府 0 1399. 216 3.68 414
2 兵庫県 0 1197. 230 3.54 591
3 千葉県 1 1124. 259 3.72 1000
4 和歌山県 0 1252 83 3.97 140
5 埼玉県 1 1147. 278 3.64 1000
6 大阪府 0 1203. 516 3.77 1000
7 奈良県 0 1169. 45 3.85 147
8 東京都 1 1283. 1165 3.67 1000
9 神奈川県 1 1239. 587 3.53 1000新しく出来たEastJapanのデータ型はなんでしょうか。
EastJapanはnumeric型ですね。もし、これをfactor型にしたい場合はどうすればよいでしょうか。それはmutate()内でEastJapanを生成した後にfactor()関数を使うだけです。
# EastJapan変数をfactor型にする
df3 <- df2 |>
mutate(EastJapan = recode(Pref,
"東京都" = 1,
"神奈川県" = 1,
"千葉県" = 1,
"埼玉県" = 1,
.default = 0),
EastJapan = factor(EastJapan, levels = c(0, 1)),
.after = Pref)
df3$EastJapan[1] 0 0 1 0 1 0 0 1 1
Levels: 0 1EastJapanがfactor型になりました。実は、recodeは再コーディングと同時にfactor化をしてくれる機能があります。ただし、recode()関数でなく、recode_factor()関数を使います。
# recode_factor()を使う方法
df3 <- df2 |>
mutate(EastJapan = recode_factor(Pref,
"東京都" = 1,
"神奈川県" = 1,
"千葉県" = 1,
"埼玉県" = 1,
.default = 0),
.after = Pref)
df3$EastJapan[1] 0 0 1 0 1 0 0 1 1
Levels: 1 0ただし、levelの順番はrecode_factor()内で定義された順番になることに注意してください。factor型のより詳細な扱いについては第16章で解説します。
15.2 行単位の操作
ここでは行単位の操作について考えたいと思います。第13.3章で使ったmyDF1を見てみましょう。
myDF1 <- data.frame(
ID = 1:5,
X1 = c(2, 4, 6, 2, 7),
Y1 = c(3, 5, 1, 1, 0),
X1D = c(4, 2, 1, 6, 9),
X2 = c(5, 5, 6, 0, 2),
Y2 = c(3, 3, 2, 3, 1),
X2D = c(8, 9, 5, 0, 1),
X3 = c(3, 0, 3, 0, 2),
Y3 = c(1, 5, 9, 1, 3),
X3D = c(9, 1, 3, 3, 8)
)
myDF1 ID X1 Y1 X1D X2 Y2 X2D X3 Y3 X3D
1 1 2 3 4 5 3 8 3 1 9
2 2 4 5 2 5 3 9 0 5 1
3 3 6 1 1 6 2 5 3 9 3
4 4 2 1 6 0 3 0 0 1 3
5 5 7 0 9 2 1 1 2 3 8ここでX1とX2とX3の平均値を計算し、X_Meanという名の変数にする場合、以下のような書き方が普通でしょう。
ID X1 Y1 X1D X2 Y2 X2D X3 Y3 X3D X_Mean
1 1 2 3 4 5 3 8 3 1 9 3.133333
2 2 4 5 2 5 3 9 0 5 1 3.133333
3 3 6 1 1 6 2 5 3 9 3 3.133333
4 4 2 1 6 0 3 0 0 1 3 3.133333
5 5 7 0 9 2 1 1 2 3 8 3.133333あら、なんかおかしくありませんか。1行目の場合、X1とX2、X3それぞれ2、5、3であり、平均値は3.333であるはずなのに3.133になりました。これは2行目以降も同じです。なぜでしょうか。
実は{dplyr}は行単位の計算が苦手です。実際、データフレームというのは既に説明したとおり、縦ベクトルを横に並べたものです。列をまたがる場合、データ型が異なる場合も多いため、そもそも使う場面も多くありません。したがって、以下のような書き方が必要でした。
ID X1 Y1 X1D X2 Y2 X2D X3 Y3 X3D X_Mean
1 1 2 3 4 5 3 8 3 1 9 3.3333333
2 2 4 5 2 5 3 9 0 5 1 3.0000000
3 3 6 1 1 6 2 5 3 9 3 5.0000000
4 4 2 1 6 0 3 0 0 1 3 0.6666667
5 5 7 0 9 2 1 1 2 3 8 3.6666667先ほどのmean(c(X1, X2, X3))は(X1列とX2列、X3列)の平均値です。X1は長さ1のベクトルではなく、その列全体を指すものです。つまり、mean(c(X1, X2, X3))はmean(c(myD1F$X1, myDF1$X2, myDF1$X3))と同じことになります。だから全て3.133という結果が得られました。ただし、後者はベクトル同士の加減乗除になるため問題ありません。実際c(1, 2, 3) + c(3, 5, 0)は同じ位置の要素同士の計算になることを既に第10.2章で説明しました。
ここでmean()関数を使う場合には全ての演算を、一行一行に分けて行う必要があります。ある一行のみに限定する場合、mean(c(X1, X2, X3))のX1などは長さ1のベクトルになるため、(X1 + X2 + X3) / 3と同じことになります。この「一行単位で処理を行う」ことを指定する関数がrowwise()関数です。これは行単位の作業を行う前に指定するだけです。
# A tibble: 5 × 11
# Rowwise:
ID X1 Y1 X1D X2 Y2 X2D X3 Y3 X3D X_Mean
<int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 1 2 3 4 5 3 8 3 1 9 3.33
2 2 4 5 2 5 3 9 0 5 1 3
3 3 6 1 1 6 2 5 3 9 3 5
4 4 2 1 6 0 3 0 0 1 3 0.667
5 5 7 0 9 2 1 1 2 3 8 3.67 これで問題なく行単位の処理ができるようになりました。今回は変数が3つのみだったので、これで問題ありませんが、変数が多くなると:やstarts_with()、num_range()などを使って変数を選択したくなります。この場合は計算する関数内にc_across()を入れます。ここではX1列からX3D列までの平均値を求めてみましょう。
# A tibble: 5 × 11
# Rowwise:
ID X1 Y1 X1D X2 Y2 X2D X3 Y3 X3D X_Mean
<int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 1 2 3 4 5 3 8 3 1 9 5.5
2 2 4 5 2 5 3 9 0 5 1 2.5
3 3 6 1 1 6 2 5 3 9 3 4.5
4 4 2 1 6 0 3 0 0 1 3 2.5
5 5 7 0 9 2 1 1 2 3 8 7.5実はrowwise()関数、2020年6月に公開されたdplyr 1.0.0で注目された関数ですが、昔の{dplyr}にもrowwise()関数はありました。ただし、{purrr}パッケージや{tidyr}パッケージのnest()関数などにより使い道がなくなりましたが、なぜか華麗に復活しました。データ分析に使うデータは基本単位は列であるため、実際にrowwise()が使われる場面は今の段階では多くないでしょう。また処理が重いことも一つのデメリットです。それでも、覚えておけば便利な関数であることには間違いありません。
15.3 データの結合
15.3.1 行の結合
まずは、複数のデータフレームまたはtibbleを縦に結合する方法について解説します。イメージとしては 図 15.1 のようなものです。
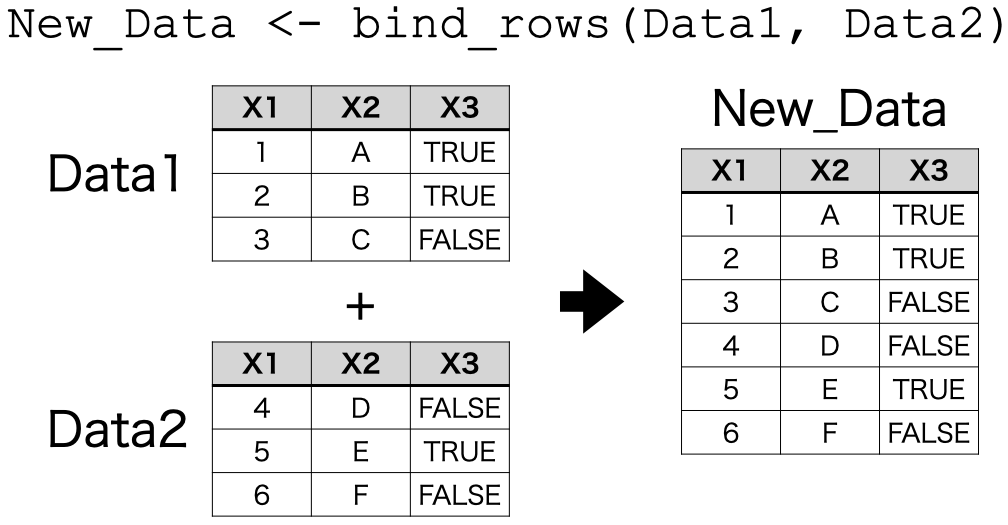
行を結合する際には{dplyr}パッケージのbind_rows()関数を使います。この関数の使い方は以下の通りです。
それでは早速実際に使ってみましょう。実習のために、4つのtibbleを作成します (tibbleでなくデータフレームでも問題ありません)。
# tibble()の代わりにdata.frame()も可
rbind_df1 <- tibble(X1 = 1:3,
X2 = c("A", "B", "C"),
X3 = c(T, T, F)) # TRUEとFALSEはTはFと省略可能
rbind_df2 <- tibble(X1 = 4:6,
X2 = c("D", "E", "F"),
X3 = c(F, T, F))
rbind_df3 <- tibble(X1 = 7:9,
X3 = c(T, T, T),
X2 = c("G", "H", "I"))
rbind_df4 <- tibble(X1 = 10:12,
X2 = c("J", "K", "L"),
X5 = c("Song", "Yanai", "Hadley"))
rbind_df1 # rbind_df1を出力# A tibble: 3 × 3
X1 X2 X3
<int> <chr> <lgl>
1 1 A TRUE
2 2 B TRUE
3 3 C FALSE# A tibble: 3 × 3
X1 X2 X3
<int> <chr> <lgl>
1 4 D FALSE
2 5 E TRUE
3 6 F FALSE# A tibble: 3 × 3
X1 X3 X2
<int> <lgl> <chr>
1 7 TRUE G
2 8 TRUE H
3 9 TRUE I # A tibble: 3 × 3
X1 X2 X5
<int> <chr> <chr>
1 10 J Song
2 11 K Yanai
3 12 L Hadleyまずは、rbind_df1とrbind_df2を結合してみます。この2つのデータは同じ変数が同じ順番で並んでいますね。
# A tibble: 6 × 3
X1 X2 X3
<int> <chr> <lgl>
1 1 A TRUE
2 2 B TRUE
3 3 C FALSE
4 4 D FALSE
5 5 E TRUE
6 6 F FALSE2つのデータが結合されたことが確認できます。それではrbind_df1とrbind_df2、rbind_df3はどうでしょうか。確かに3つのデータは同じ変数を持ちますが、rbind_df3は変数の順番がX1、X3、X2になっています。このまま結合するとエラーが出るでしょうか。とりあえず、やってみます。
# A tibble: 9 × 3
X1 X2 X3
<int> <chr> <lgl>
1 1 A TRUE
2 2 B TRUE
3 3 C FALSE
4 4 D FALSE
5 5 E TRUE
6 6 F FALSE
7 7 G TRUE
8 8 H TRUE
9 9 I TRUE このように変数の順番が異なっても、先に指定したデータの変数順で問題なく結合できました。これまでの作業は{dplyr}パッケージのbind_rows()を使わずに、R内蔵関数のrbind()でも同じやり方でできます。bind_rows()の特徴は、変数名が一致しない場合、つまり今回の例だとrbind_df4が含まれる場合です。rbind_df1からrbind_df3までは順番が違ってもX1、X2、X3変数で構成されていました。一方、rbind_dr4にはX3がなく、新たにX4という変数があります。これをrbind()関数で結合するとエラーが出力されます。
Error in match.names(clabs, names(xi)): names do not match previous names一方、bind_rows()はどうでしょうか。
# A tibble: 12 × 4
X1 X2 X3 X5
<int> <chr> <lgl> <chr>
1 1 A TRUE <NA>
2 2 B TRUE <NA>
3 3 C FALSE <NA>
4 4 D FALSE <NA>
5 5 E TRUE <NA>
6 6 F FALSE <NA>
7 7 G TRUE <NA>
8 8 H TRUE <NA>
9 9 I TRUE <NA>
10 10 J NA Song
11 11 K NA Yanai
12 12 L NA HadleyX1からX4まで全ての列が生成され、元のデータにはなかった列に関してはNAで埋められています。
他にもbind_rows()には.idという便利な引数があります。これは結合の際に新しい列を追加し、結合前のデータごとの固有の値を割り当ててくれます。たとえば、rbind_df1とrbind_df2を結合し、それぞれ"Data1"、"Data2"という値を割り当てるとしましょう。この場合、2つの表をlist()関数でまとめ、.idには新しく追加される列名を指定します。
# A tibble: 6 × 4
Data X1 X2 X3
<chr> <int> <chr> <lgl>
1 Data1 1 A TRUE
2 Data1 2 B TRUE
3 Data1 3 C FALSE
4 Data2 4 D FALSE
5 Data2 5 E TRUE
6 Data2 6 F FALSE予めリストを作っておいて、それをbind_rows()に渡すこともできます。
# A tibble: 6 × 4
Data X1 X2 X3
<chr> <int> <chr> <lgl>
1 Data1 1 A TRUE
2 Data1 2 B TRUE
3 Data1 3 C FALSE
4 Data2 4 D FALSE
5 Data2 5 E TRUE
6 Data2 6 F FALSEこのようにbind_rows()はrbind()よりも便利です。しかし、bind_rows()の完全勝利かというと、そうとは限りません。自分で架空した複数のデータフレーム、またはtibbleを結合する際、「このデータは全て同じ変数を持っているはず」と事前に分かっているならrbind()の方が効果的です。なぜなら、変数名が異なる場合、エラーが出力されるからです。bind_rows()を使うと、コーディングミスなどにより、列名の相違がある場合でも結合してくれてしまうので、分析の結果を歪ませる可能性があります。
15.3.2 列の結合
実はデータ分析においてデータの結合といえば、列の結合が一般的です。これは 図 15.2 のような操作を意味します。
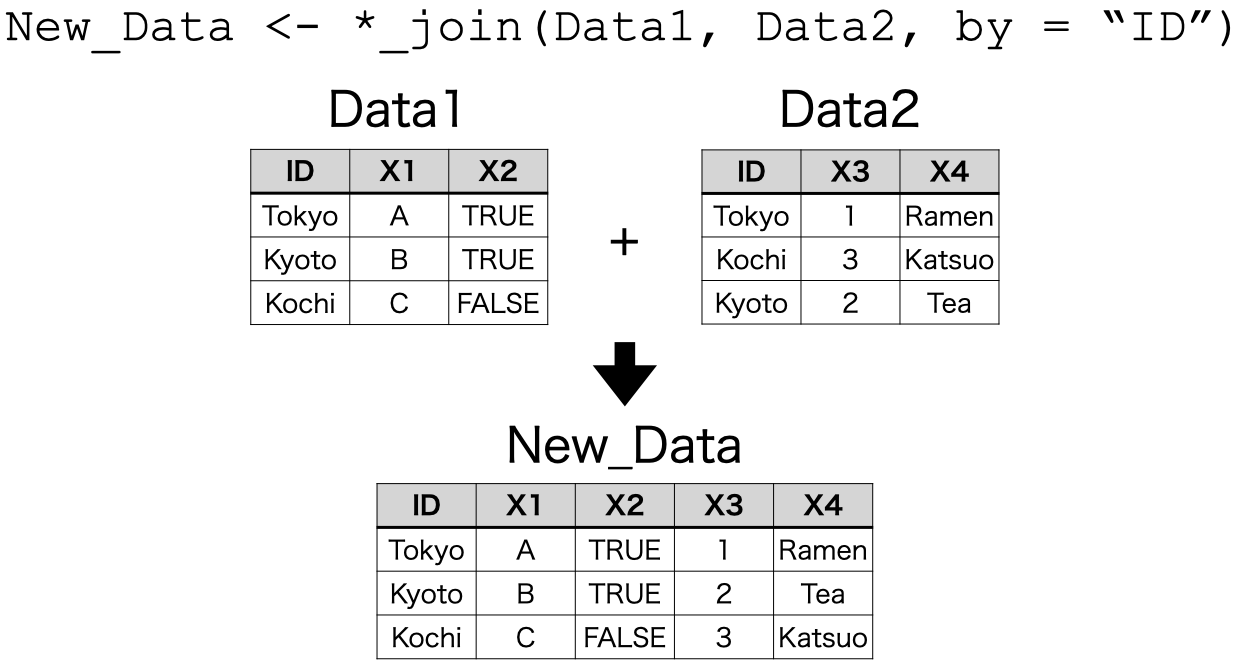
まずは、本章で作成したdf2をもう一回作ってみます。
df2 <- df |>
group_by(Pref) |>
summarise(Budget_Mean = mean(Budget, na.rm = TRUE),
ScoreN_Sum = sum(ScoreN, na.rm = TRUE),
Score_Mean = mean(Score, na.rm = TRUE),
N = n(),
.groups = "drop")
df2# A tibble: 9 × 5
Pref Budget_Mean ScoreN_Sum Score_Mean N
<chr> <dbl> <dbl> <dbl> <int>
1 京都府 1399. 216 3.68 414
2 兵庫県 1197. 230 3.54 591
3 千葉県 1124. 259 3.72 1000
4 和歌山県 1252 83 3.97 140
5 埼玉県 1147. 278 3.64 1000
6 大阪府 1203. 516 3.77 1000
7 奈良県 1169. 45 3.85 147
8 東京都 1283. 1165 3.67 1000
9 神奈川県 1239. 587 3.53 1000ラーメン屋の店舗ですが、たしかにデータには埼玉、東京、大阪などは1000店舗しか入っておりません。実はもっと多いですが、ぐるなびAPIの仕様上、最大1000店舗しか情報取得が出来ないからです。ここに実際の店舗数が入っている新しいデータセット、Ramen2.csvがあります。これを読み込み、df3という名で格納しましょう。
Rows: 47 Columns: 15
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (1): Pref
dbl (12): RamenN, Turnout, LDP, CDP, DPFP, Komei, JIP, JCP, SDP, Reiwa, NHK,...
num (2): Pop, Area
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.# A tibble: 47 × 15
Pref Pop Area RamenN Turnout LDP CDP DPFP Komei JIP JCP SDP
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 北海道 5.38e6 83424. 1454 53.8 32.3 20.8 6.65 11.7 7.78 11.6 1.31
2 青森県 1.31e6 9646. 336 42.9 39.8 22.0 7.24 11.3 3.4 8.31 2.36
3 岩手県 1.28e6 15275. 285 56.5 35.5 17.8 12.5 8.22 4.36 10.4 3.83
4 宮城県 2.33e6 7282. 557 51.2 39.6 17.8 9.02 11.1 4.6 7.89 2.1
5 秋田県 1.02e6 11638. 301 56.3 44.5 13.5 8.64 10.6 4.48 8.09 3.77
6 山形県 1.12e6 9323. 512 60.7 45.2 14.9 7.37 9.87 4.28 6.51 5.08
7 福島県 1.91e6 13784. 550 52.4 38.2 13.6 12.1 12.8 5.31 7.99 3.01
8 茨城県 2.92e6 6097. 663 45.0 39.3 15.2 7.15 15.1 6.73 7.73 1.46
9 栃木県 1.97e6 6408. 595 44.1 40.3 18.9 9.94 12.8 4.9 5.04 1.03
10 群馬県 1.97e6 6362. 488 48.2 40.6 16.4 9.76 12.4 4.67 7.58 1.87
# ℹ 37 more rows
# ℹ 3 more variables: Reiwa <dbl>, NHK <dbl>, HRP <dbl>Ramen2.csvの詳細
| 変数名 | 説明 |
|---|---|
Pref |
都道府県名 |
Pop |
日本人人口 (2015年国勢調査) |
Area |
面積 (2015年国勢調査) |
RamenN |
ぐるなびに登録されたラーメン屋の店舗数 |
Turnout |
2019年参院選: 投票率 (比例) |
LDP |
2019年参院選: 自民党の得票率 (比例) |
CDP |
2019年参院選: 立憲民主党の得票率 (比例) |
DPFP |
2019年参院選: 国民民主党の得票率 (比例) |
Komei |
2019年参院選: 公明党の得票率 (比例) |
JIP |
2019年参院選: 日本維新の会の得票率 (比例) |
JCP |
2019年参院選: 日本共産党の得票率 (比例) |
SDP |
2019年参院選: 社会民主党の得票率 (比例) |
Reiwa |
2019年参院選: れいわ新選組の得票率 (比例) |
NHK |
2019年参院選: NHKから国民を守る党の得票率 (比例) |
HRP |
2019年参院選: 幸福実現党の得票率 (比例) |
本データは都道府県ごとの人口、面積、ぐるなびに登録されたラーメン屋の店舗数、2019年参議院議員通常選挙の結果が格納されています。人口と面積は2015年国勢調査、ぐるなびの情報は2020年6月時点での情報です。
df2にデータ上の店舗数ではなく、実際の店舗数を新しい列として追加したい場合はどうすれば良いでしょうか。簡単な方法としてはdf3から情報を取得し、それを自分で入れる方法です。
- 1
- df2のPrefベクトルの要素と一致するものに絞る
- 2
- 都道府県名とラーメン屋の店舗数のみ抽出
# A tibble: 9 × 2
Pref RamenN
<chr> <dbl>
1 埼玉県 1106
2 千葉県 1098
3 東京都 3220
4 神奈川県 1254
5 京都府 415
6 大阪府 1325
7 兵庫県 591
8 奈良県 147
9 和歌山県 140そして、この情報をdf2$RamenN <- c(415, 1106, 1254, ...)のように追加すればいいですね。
しかし、このような方法は非効率的です。そもそもdf3から得られた結果の順番とdf2の順番も一致しないので、一々対照しながらベクトルを作ることになります。ここで登場する関数が{dplyr}の*_join()関数群です。この関数群には4つの関数が含まれており、以下のような使い方になります。
# 新しいデータ名ではなく、データ1またはデータ2の名前に格納すると上書きとなる
# 1. データ1を基準に結合
新しいデータ名 <- left_join(データ1, データ2, by = "共通変数名")
# 2. データ2を基準に結合
新しいデータ名 <- right_join(データ1, データ2, by = "共通変数名")
# 3. データ1とデータ2両方に共通するケースのみ結合
新しいデータ名 <- inner_join(データ1, データ2, by = "共通変数名")
# 4. データ1とデータ2、どれかに存在するケースを結合
新しいデータ名 <- full_join(データ1, データ2, by = "共通変数名")4つの関数の違いについて説明する前に、by引数について話したいと思います。これは主にキー (key)変数と呼ばれる変数で、それぞれのデータに同じ名前の変数がある必要があります。df2とdf3だとそれがPref変数です。どの*_join()関数でも、Prefの値が同じもの同士を結合することになります。
データのキー変数名が異なる場合もあります。たとえば、データ1の都道府県名はPrefという列に、データ2の都道府県名はPrefectureという列になっている場合、by = "Pref"でなく、by = c("データ1のキー変数名" = "データ2のキー変数名")、つまり、by = c("Pref" = "Prefecture")と指定します。
それでは、df3から都道府県名とラーメン屋の店舗数だけ抽出し、df4として格納しておきます。
# A tibble: 47 × 2
Pref RamenN
<chr> <dbl>
1 北海道 1454
2 青森県 336
3 岩手県 285
4 宮城県 557
5 秋田県 301
6 山形県 512
7 福島県 550
8 茨城県 663
9 栃木県 595
10 群馬県 488
# ℹ 37 more rowsこれから共通変数名の値をキー (key)と呼びます。今回の例だとPrefがdf2とdf4のキー変数であり、その値である"東京都"、"北海道"などがキーです。
まずは、inner_join()の仕組みについて考えます。これはdf2とdf4に共通するキーを持つケースのみ結合する関数です。df4には"北海道"というキーがありますが、df2にはありません。したがって、キーが"北海道"のケースは結合から除外されます。これをイメージにしたものが 図 15.3 です2。それぞれ3 \(\times\) 2 (3行2列)のデータですが、キーが一致するケースは2つしかないため、結合後のデータは3 \(\times\) 2となります。
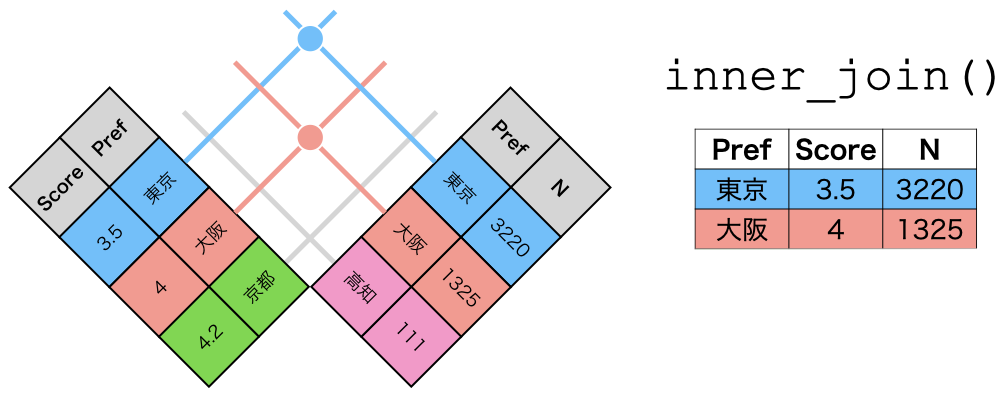
inner_join()の仕組み
実際にやってみましょう。
# A tibble: 9 × 6
Pref Budget_Mean ScoreN_Sum Score_Mean N RamenN
<chr> <dbl> <dbl> <dbl> <int> <dbl>
1 京都府 1399. 216 3.68 414 415
2 兵庫県 1197. 230 3.54 591 591
3 千葉県 1124. 259 3.72 1000 1098
4 和歌山県 1252 83 3.97 140 140
5 埼玉県 1147. 278 3.64 1000 1106
6 大阪府 1203. 516 3.77 1000 1325
7 奈良県 1169. 45 3.85 147 147
8 東京都 1283. 1165 3.67 1000 3220
9 神奈川県 1239. 587 3.53 1000 1254共通するキーは9つのみであり、結果として返されたデータの大きさも9 \(\times\) 6です。df2に足されたdf4は2列のデータですが、キー変数であるPrefは共通するため、1列のみ足されました。キー変数を両方残す場合はkeep = TRUE引数を追加してください。
一方、full_join()は、すべてのキーに対して結合を行います ( 図 15.4 )。たとえば、df2には"北海道"というキーがありません。それでも新しく出来上がるデータには北海道の列が追加されます。ただし、道内店舗の平均予算、口コミ数などの情報はないため、欠損値が代入されます。
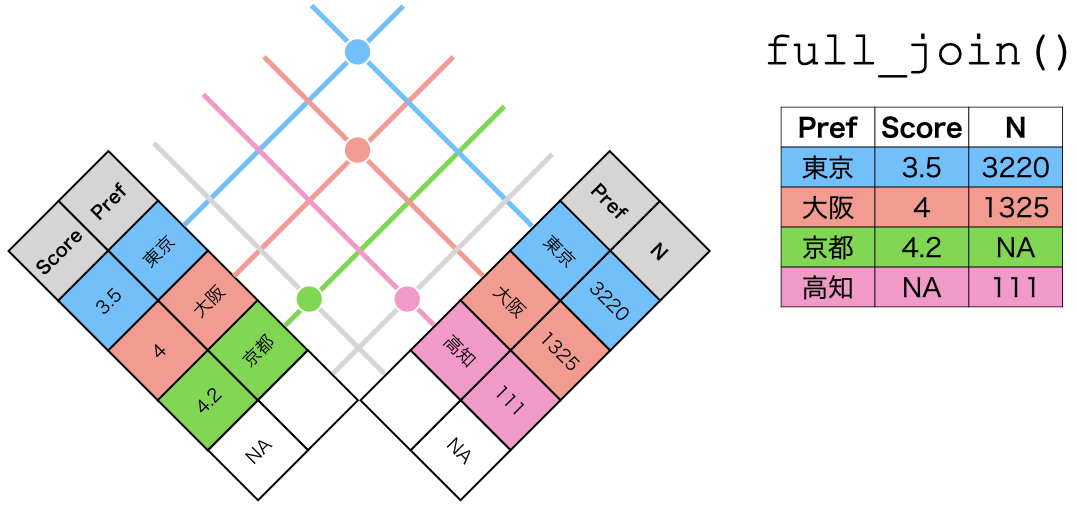
full_join()の仕組み
それでは実際、結果を確認してみましょう。今回は結合後、RamenNが大きい順で出力します。
# A tibble: 47 × 6
Pref Budget_Mean ScoreN_Sum Score_Mean N RamenN
<chr> <dbl> <dbl> <dbl> <int> <dbl>
1 東京都 1283. 1165 3.67 1000 3220
2 北海道 NA NA NA NA 1454
3 大阪府 1203. 516 3.77 1000 1325
4 愛知県 NA NA NA NA 1255
5 神奈川県 1239. 587 3.53 1000 1254
6 埼玉県 1147. 278 3.64 1000 1106
7 千葉県 1124. 259 3.72 1000 1098
8 福岡県 NA NA NA NA 985
9 新潟県 NA NA NA NA 705
10 静岡県 NA NA NA NA 679
# ℹ 37 more rowsdf2にはなかった北海道や愛知県などの行ができました。そして、df2にはない情報はすべて欠損値 (NA)となりました。
続いて、left_join()ですが、これは先に指定したデータに存在するキーのみで結合を行います ( 図 15.5 )。今回はdf2が先に指定されていますが、df2のキーはdf4のキーの部分集合であるため、inner_join()と同じ結果が得られます。
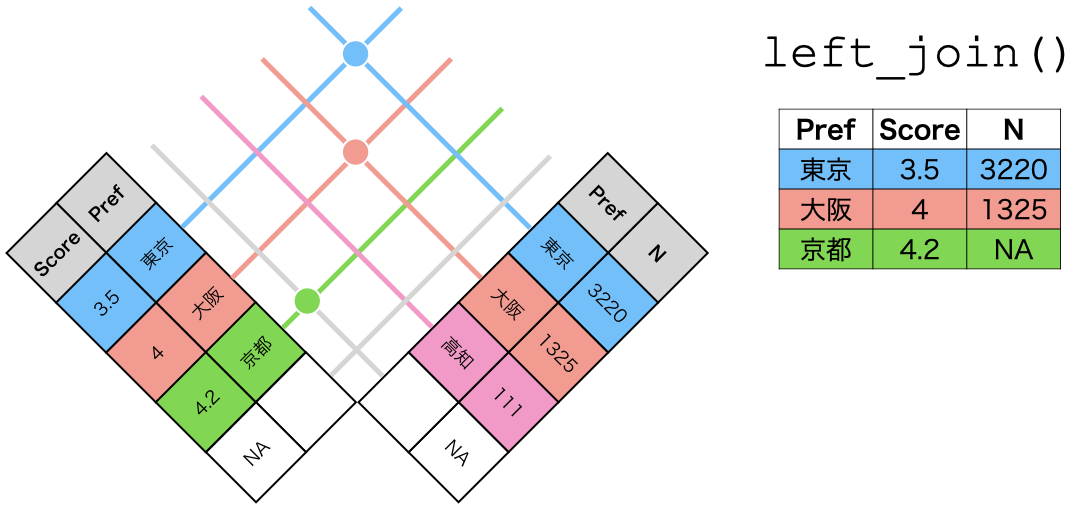
left_join()の仕組み
一方、right_join()はleft_join()と逆の関数であり、後に指定したデータに存在するキーを基準に結合を行います( 図 15.6 )。後に指定されたdf4のキーはdf2のキーを完全に含むので、full_join()と同じ結果が得られます。
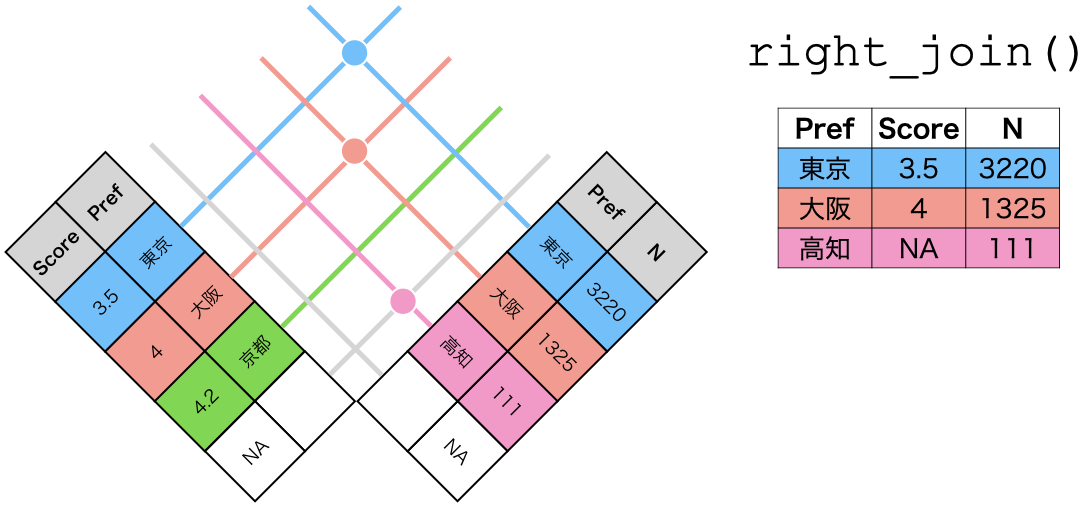
right_join()の仕組み
これからはdf2とdf4を結合することになりますが、この2つのtibbleの大きさが異なります。df2は9つの都府県のみであるに対し、df4は47都道府県全てのデータが入っているからです。
ここまではキー変数が一つである場合についてのみ考えましたが、複数のキー変数が必要な場合もあります。たとえば、市区町村の人口・面積データと市区町村の投票率データを結合するとします。各自治体に与えられている「全国地方公共団体コード」が両データに含まれている場合は、このコードをキー変数として使えば問題ありませんが、市区町村名をキー変数として使わざる得ないケースもあるでしょう。しかし、キー変数が複数ある場合もあります。たとえば、府中市は東京都と広島県にありますし、太子町は大阪府と兵庫県にあります。この場合、市区町村名のみでケースをマッチングすると、重複されてマッチングされる恐れがあります。この場合はキー変数を増やすことで対処できます。たとえば、同じ都道府県なら同じ市区町村は存在しないでしょう3。キー変数を複数指定する方法は簡単です。たとえば、市区町村名変数がMunip、都道府県名変数がPrefならby = c("Munip", "Pref")と指定するだけです。
最後に、キー変数以外の変数名が重複する場合について考えましょう。これはパネルデータを結合する時によく直面する問題です。同じ回答者に2回の調査を行った場合、回答者のIDでデータを結合することになります。ただし、それぞれのデータにおいて回答者の性別に関する変数がF1という名前の場合、どうなるでしょうか。同じデータの同じ名前の変数が複数あると、非常に扱いにくくなります。実際の結果を見てみましょう。
Wave1_df <- tibble(ID = c(1, 2, 3, 4, 5),
F1 = c(1, 1, 0, 0, 1),
F2 = c(18, 77, 37, 50, 41),
Q1 = c(1, 5, 2, 2, 3))
Wave2_df <- tibble(ID = c(1, 3, 4, 6, 7),
F1 = c(1, 0, 0, 0, 1),
F2 = c(18, 37, 50, 20, 62),
Q1 = c(1, 2, 2, 5, 4))
full_join(Wave1_df, Wave2_df, by = "ID")# A tibble: 7 × 7
ID F1.x F2.x Q1.x F1.y F2.y Q1.y
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 1 1 18 1 1 18 1
2 2 1 77 5 NA NA NA
3 3 0 37 2 0 37 2
4 4 0 50 2 0 50 2
5 5 1 41 3 NA NA NA
6 6 NA NA NA 0 20 5
7 7 NA NA NA 1 62 4それぞれの変数名の後に.xと.yが付きます。この接尾辞(suffix)はsuffix引数を指定することで、分析側からカスタマイズ可能です。たとえば、接尾辞を_W1、_W2にしたい場合は
# A tibble: 7 × 7
ID F1_W1 F2_W1 Q1_W1 F1_W2 F2_W2 Q1_W2
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 1 1 18 1 1 18 1
2 2 1 77 5 NA NA NA
3 3 0 37 2 0 37 2
4 4 0 50 2 0 50 2
5 5 1 41 3 NA NA NA
6 6 NA NA NA 0 20 5
7 7 NA NA NA 1 62 4のように、データ1とデータ2それぞれの接尾辞を指定するだけです。
15.3.3 キー変数のない列の結合
*_join()関数群はby引数でキー変数を指定する必要がある。一方、キー変数がなく、単純に2つの表を横に結合するケースも稀ながらあろう。以下のmy_data1とmy_data2は共通するキー変数がなく、単純に横に並べれば良い。
この場合はbind_cols()関数を使用する。()内には結合するtibble、またはdata.frameのオブジェクト名を入れるだけだ(3つ以上でも良い)。
# A tibble: 3 × 5
ID X1 X2 X3 X4
<chr> <chr> <lgl> <dbl> <chr>
1 Tokyo A TRUE 1 Ramen
2 Osaka B TRUE 3 Sauce
3 Kochi C FALSE 2 Katsuo ちなみにbind_cols()と同じ機能を持つcbind()関数もあり、こちらはR内蔵関数だ。違いがあるとすれば、cbind()の場合、必ずdata.frameクラスで結果を返す点だ。my_data1とmy_data2はtibbleクラスを持つが、cbind()で結合するとdata.frameクラスになる。一方、bind_cols()の場合、先に指定したオブジェクトのクラスと同じクラスが返される。